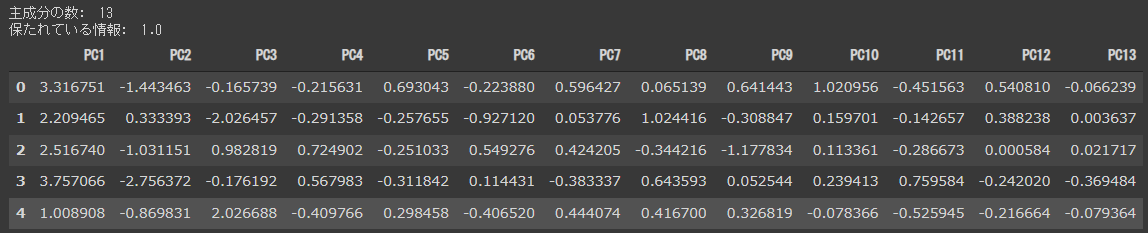
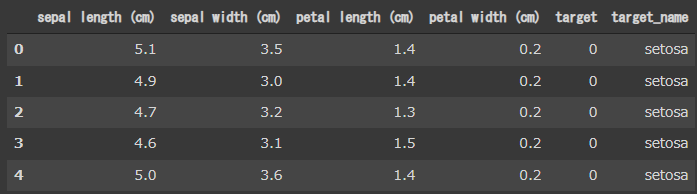
n_neighborsは、UMAPの重要なパラメータです。
n_neighborsを大きくするとマクロな構造を反映し、小さくするとミクロな構造を結果に反映することができます。
デフォルト値は15で、2~100の間の値を選択することが推奨されています。
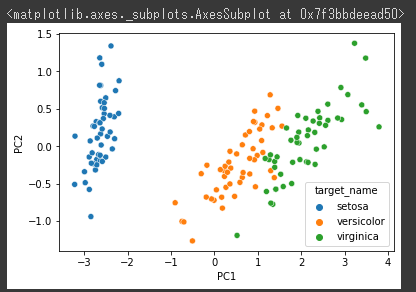
最適n_neighborsを探索(2次元)
最適なn_neighborsを探索する関数を定義します。
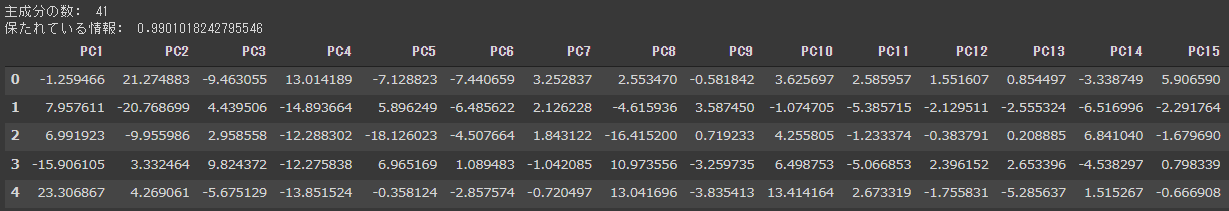
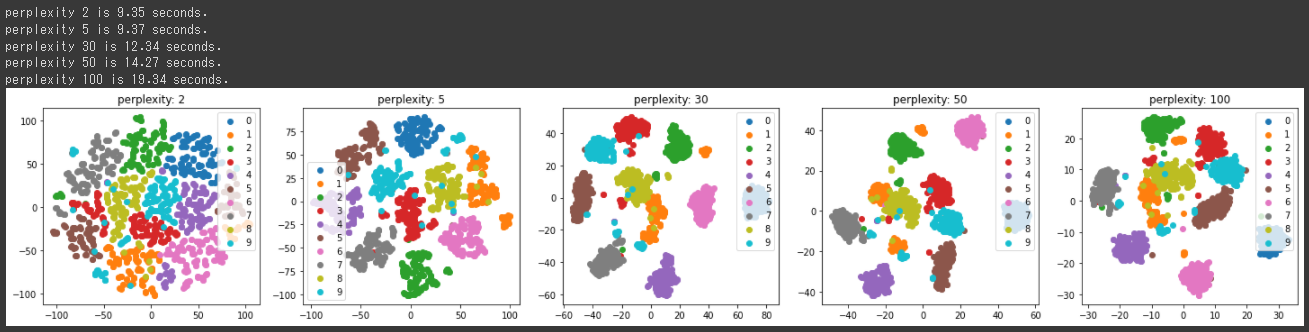
n_neighborsを2, 15, 30, 50, 100と設定してUMAPを実行し、結果を2次元に可視化します。
[Google Colaboratory]
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
| def create_2d_umap(target_X, y, y_labels, n_neighbors_list= [2, 15, 30, 50, 100]):
fig, axes = plt.subplots(nrows=1, ncols=len(n_neighbors_list),figsize=(5*len(n_neighbors_list), 4))
for i, (ax, n_neighbors) in enumerate(zip(axes.flatten(), n_neighbors_list)):
start_time = time.time()
mapper = umap.UMAP(n_components=2, random_state=0, n_neighbors=n_neighbors)
Y = mapper.fit_transform(target_X)
for each_label in y_labels:
c_plot_bool = y == each_label
ax.scatter(Y[c_plot_bool, 0], Y[c_plot_bool, 1], label="{}".format(each_label))
end_time = time.time()
ax.legend(loc="upper right")
ax.set_title("n_neighbors: {}".format(n_neighbors))
print("n_neighbors {} is {:.2f} seconds.".format(n_neighbors, end_time - start_time))
plt.show()
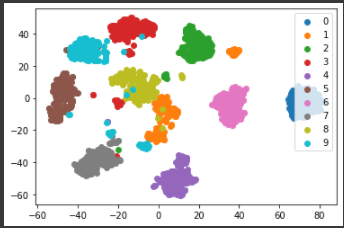
create_2d_umap(digits.data, digits.target, digits.target_names)
|
[実行結果]
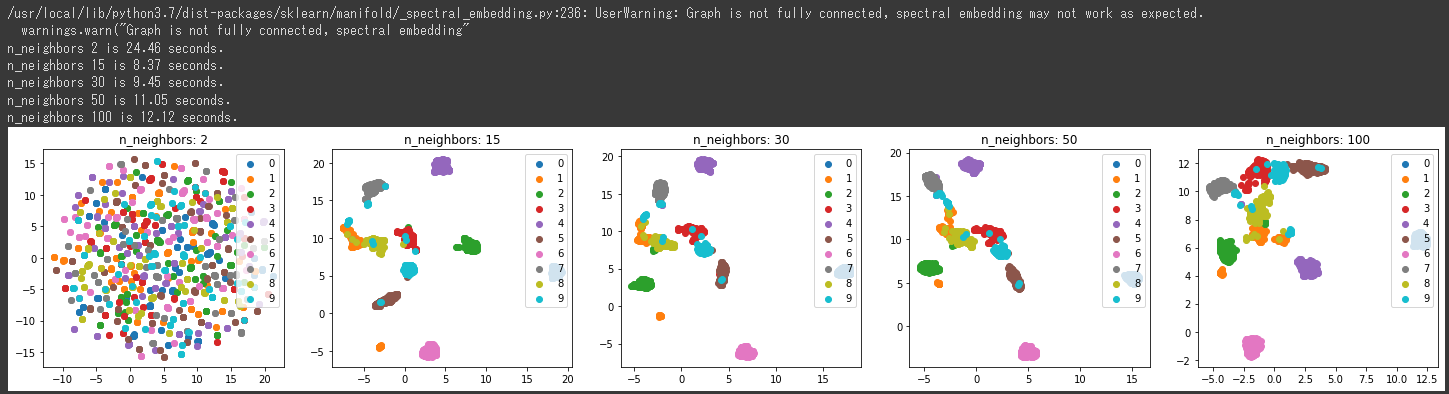
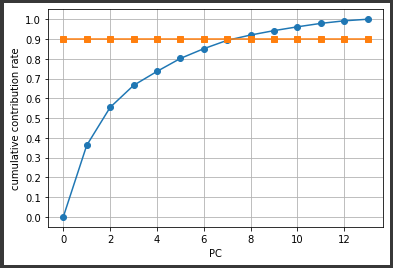
n_neighborsの値によって、結果がかなり変化することが分かります。
上記の2次元グラフからは15, 30あたりが良い結果になっているようです。
最適n_neighborsを探索(3次元)
今度は、3次元で最適なn_neighborsを探索する関数を定義します。
[Google Colaboratory]
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
| def create_3d_umap(target_X, y, y_labels, n_neighbors_list= [2, 15, 30, 50, 100]):
fig = plt.figure(figsize=(5*len(n_neighbors_list),4))
for i, n_neighbors in enumerate(n_neighbors_list):
ax = fig.add_subplot(1, len(n_neighbors_list), i+1, projection="3d")
start_time = time.time()
mapper = umap.UMAP(n_components=3, random_state=0, n_neighbors=n_neighbors)
Y = mapper.fit_transform(target_X)
for each_label in y_labels:
c_plot_bool = y == each_label
ax.scatter(Y[c_plot_bool, 0], Y[c_plot_bool, 1], label="{}".format(each_label))
end_time = time.time()
ax.legend(loc="upper right")
ax.set_title("n_neighbors_list: {}".format(n_neighbors))
print("n_neighbors_list {} is {:.2f} seconds.".format(n_neighbors, end_time - start_time))
plt.show()
create_3d_umap(digits.data, digits.target, digits.target_names)
|
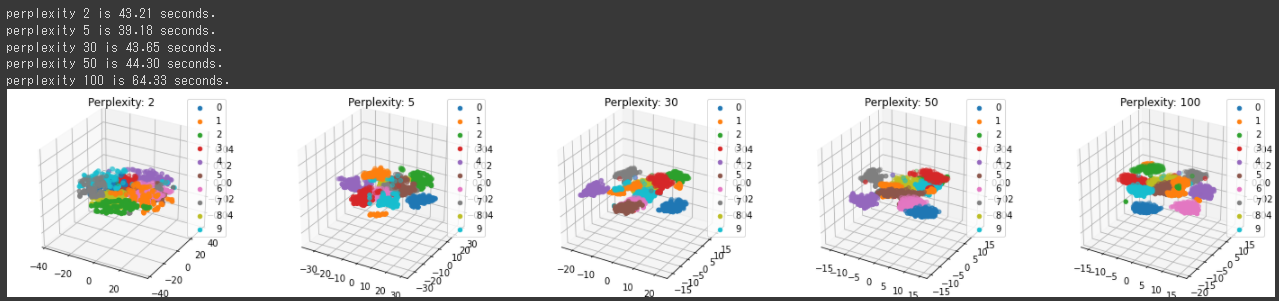
projectionパラメータに“3d”を設定(4行目)し、3次元のグラフを表示します。
また、n_componentsパラメータを2から3に変更しています。(6行目)
[実行結果]
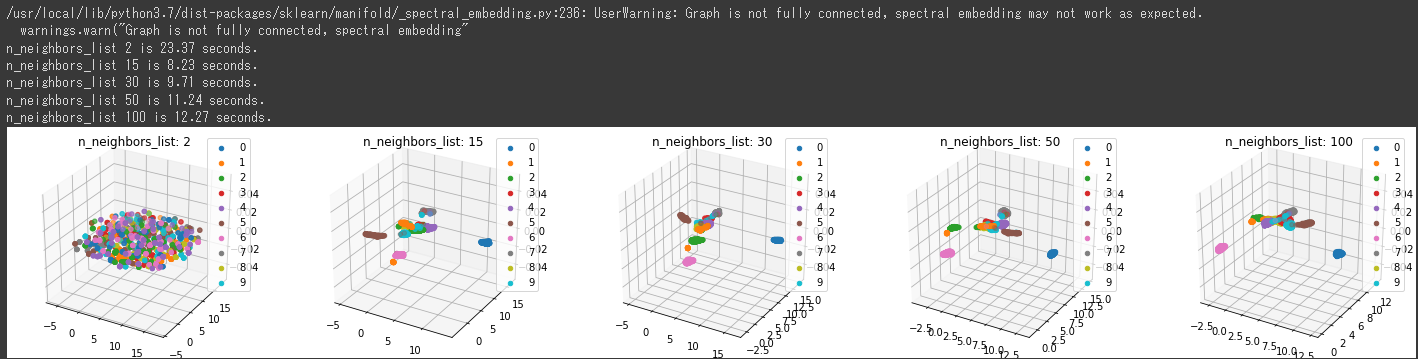
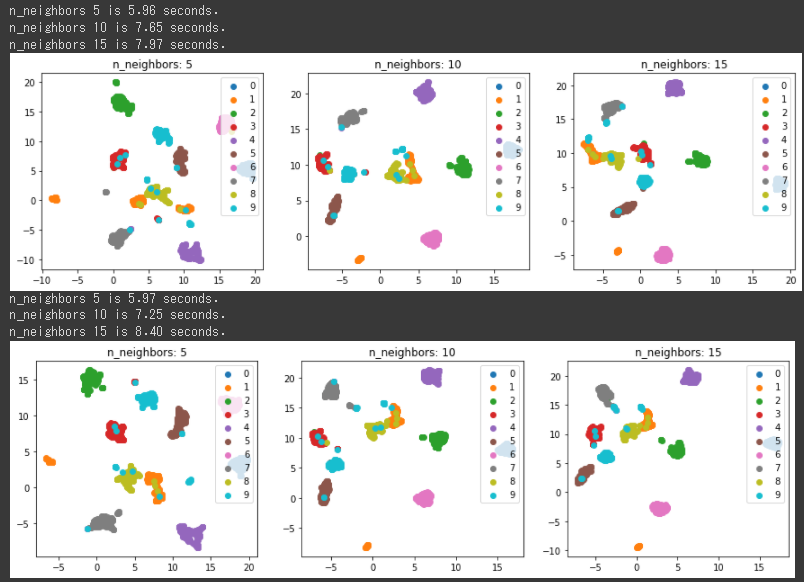
上記の3次元グラフからは15, 30あたりでの分類結果がよさそうです。
もう少しn_neighborsを変化させて、再度最適値を調べます。
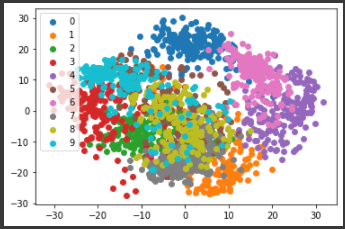
n_neighborsに[10 , 15, 20, 25, 30]を設定して実行します。
[Google Colaboratory]
1
| create_3d_umap(digits.data, digits.target, digits.target_names, [10 , 15, 20, 25, 30])
|
[実行結果]
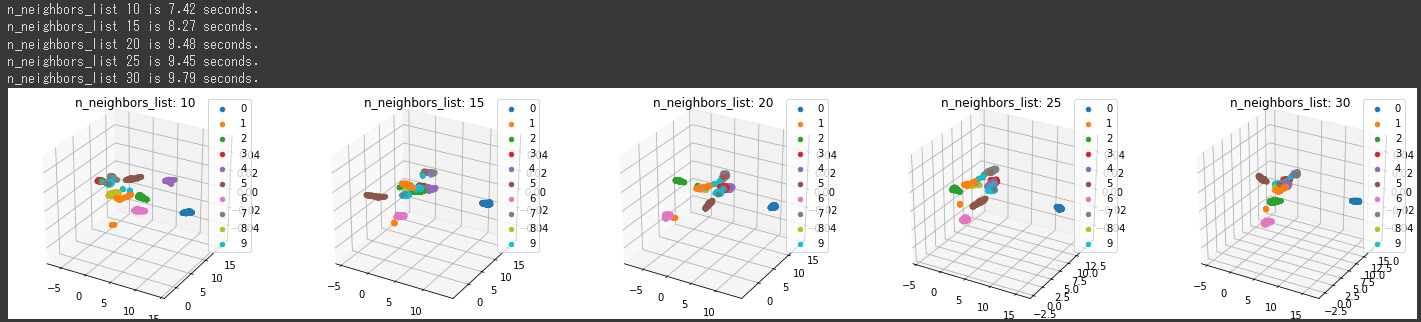
n_neighborsが10のときに最もうまく分類されています。
UMAPにおいても、n_neighborsの最適値がいくつかというのはデータセットによって違いますので、このように関数化して設定値を変更させながら比較すると最適な設定値を確認しやすくなります。