Dash DAQ
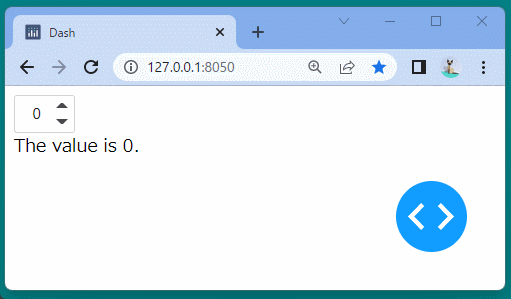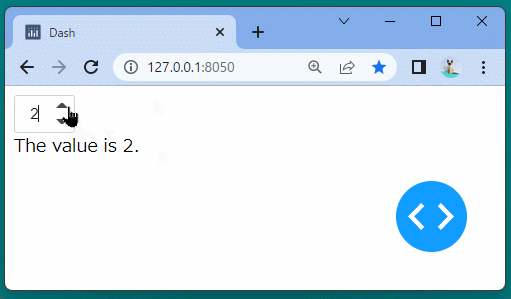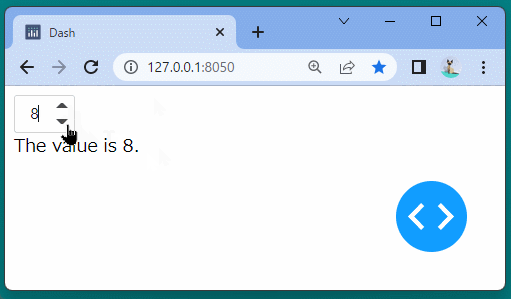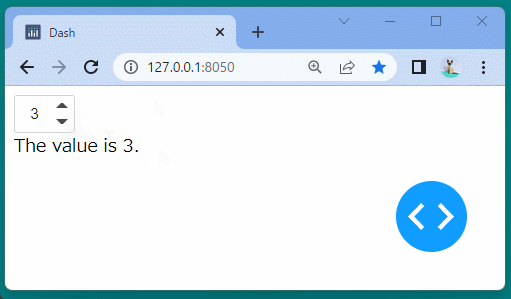
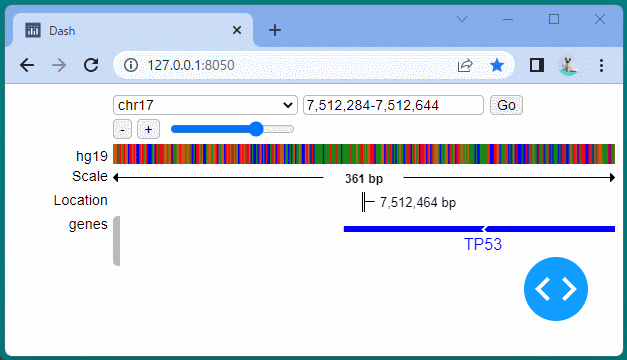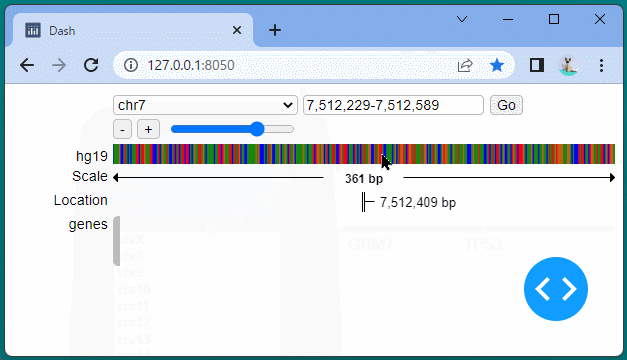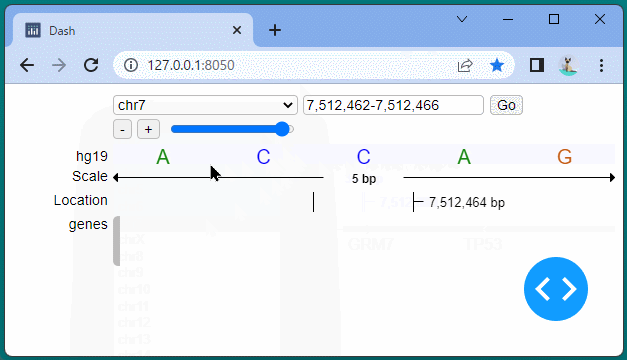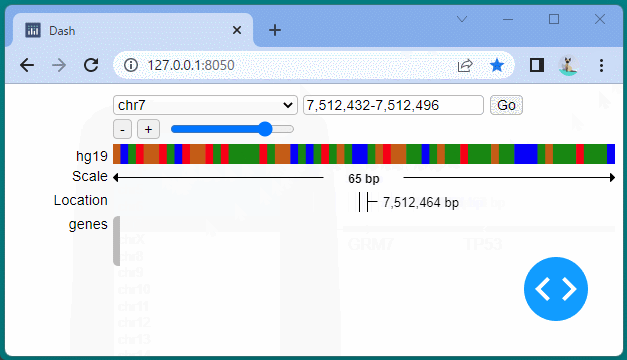
Dash DAQでは、データの収集・管理を行うコンポーネントを提供しています。
スイッチや計器に似た可視化ツールが用意されており、データを変化させる管理UIを作成できます。
Dash Enterprise - https://dash.plotly.com/dash-daq
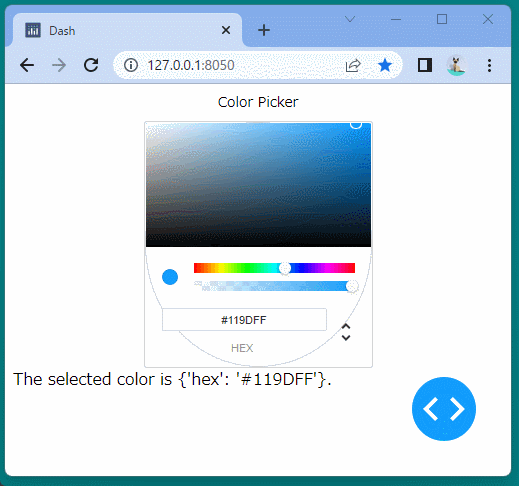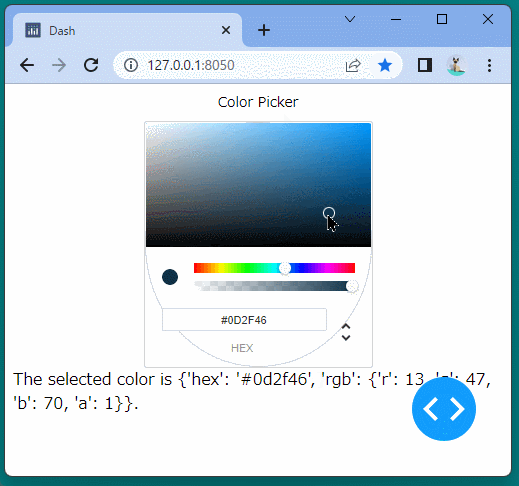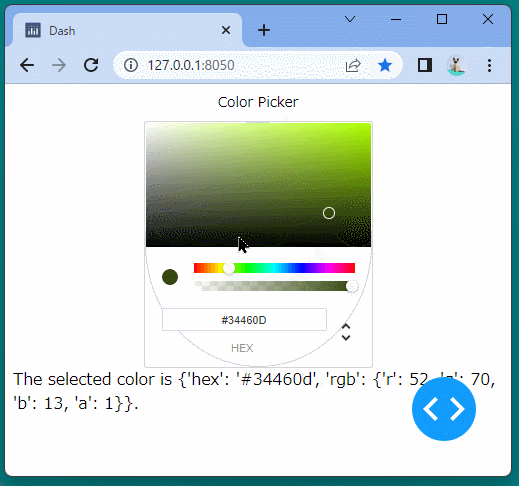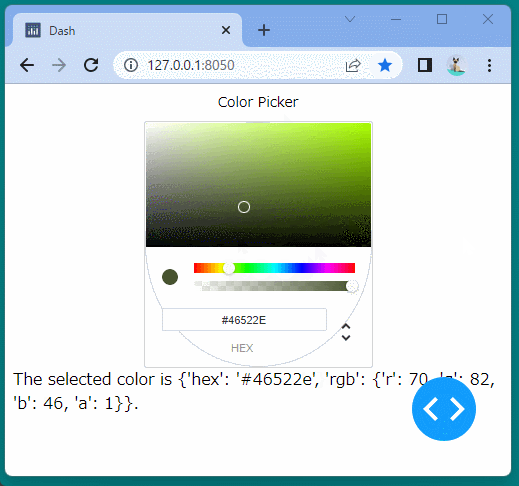
Indicatorコンポーネント
Indicatorコンポーネントを使うと、ブール型のインジケーターを表示することができます。
[ソースコード]
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
| import dash
from dash.dependencies import Input, Output
import dash_daq as daq
import dash_html_components as html
app = dash.Dash(__name__)
app.layout = html.Div([
daq.Indicator(
id='our-indicator',
label="Default"
),
html.Button(
'On/Off',
id='our-indicator-button',
n_clicks=0
)
])
@app.callback(
Output('our-indicator', 'value'),
Input('our-indicator-button', 'n_clicks')
)
def update_output(value):
return True if value % 2 == 0 else False
if __name__ == '__main__':
app.run_server(debug=True)
|
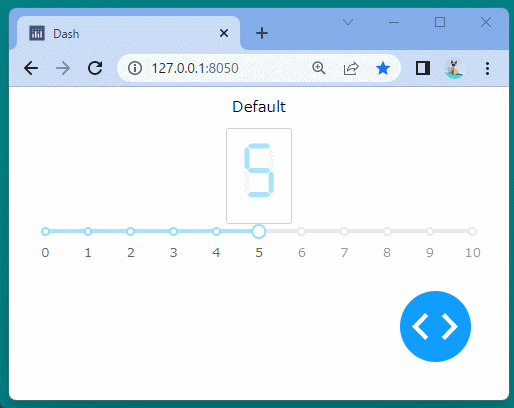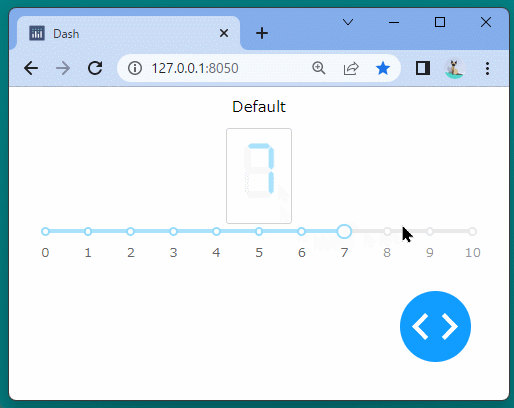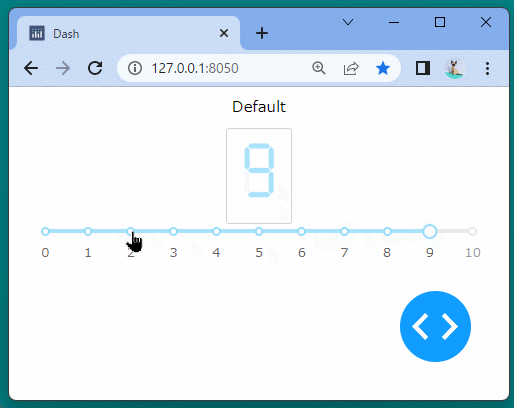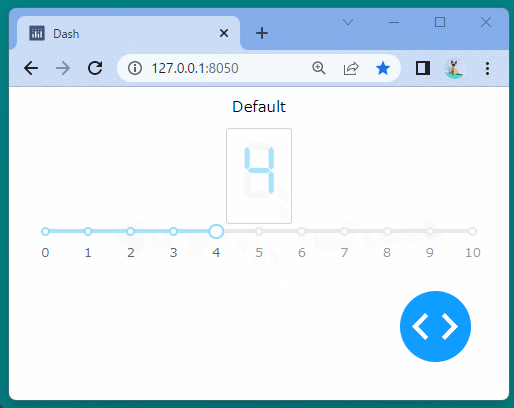
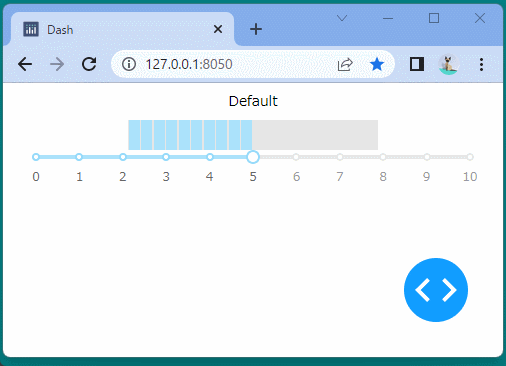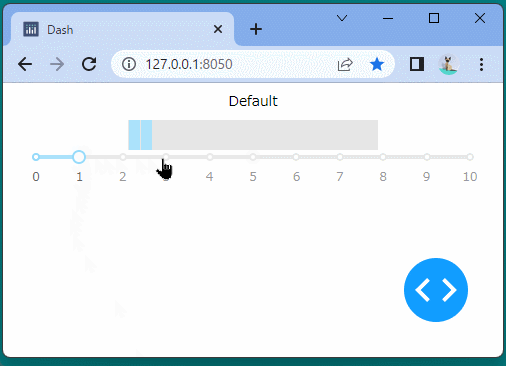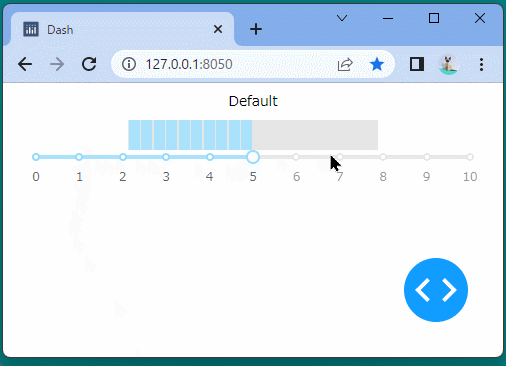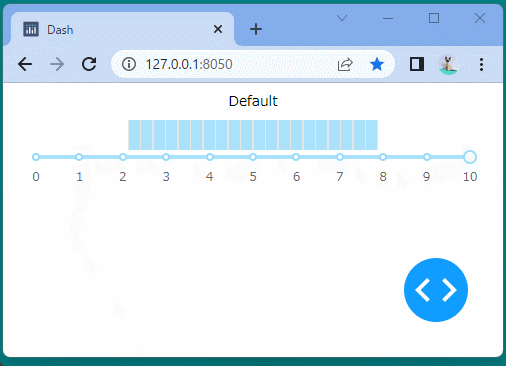
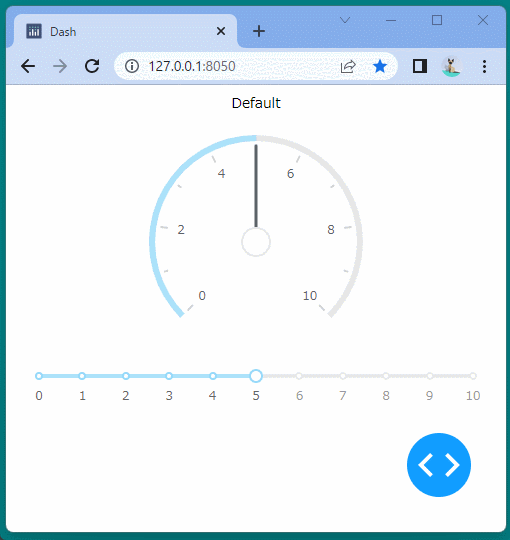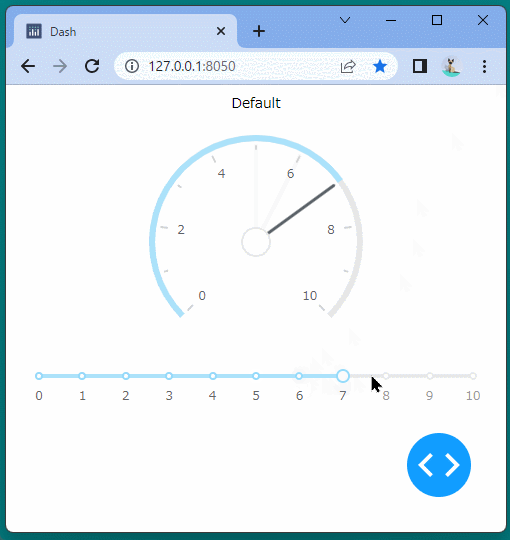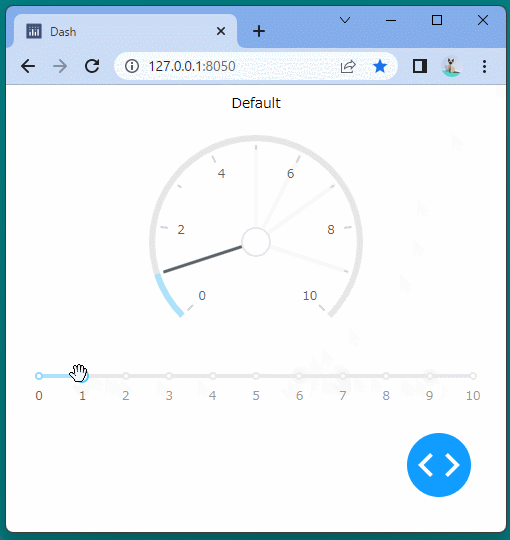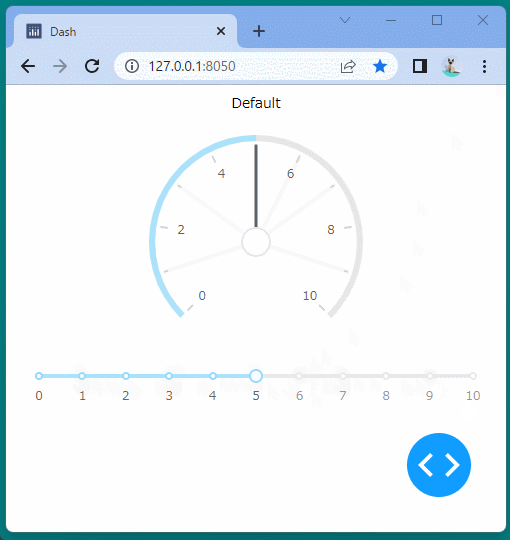
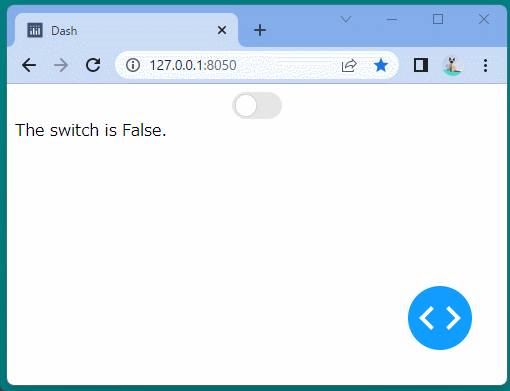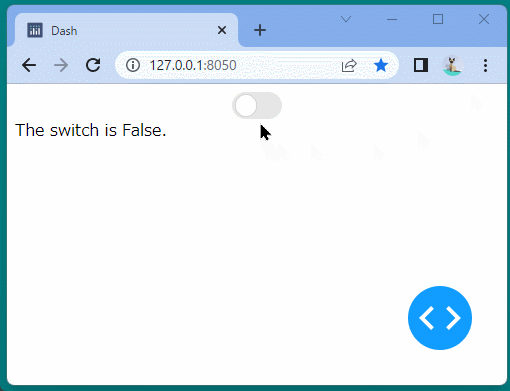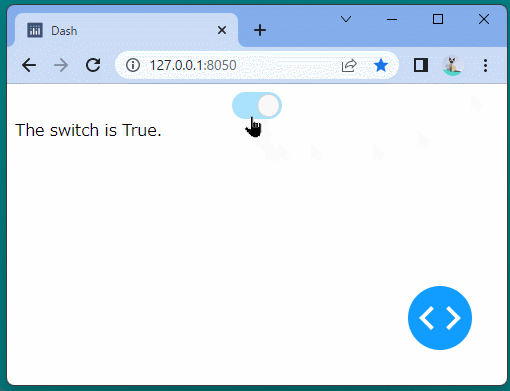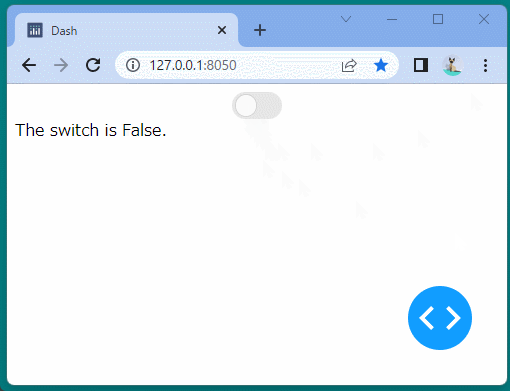
[ブラウザで表示]
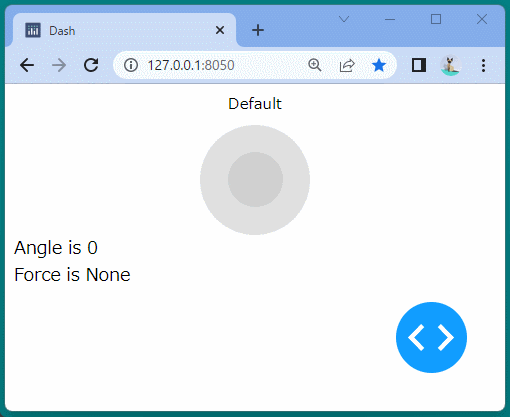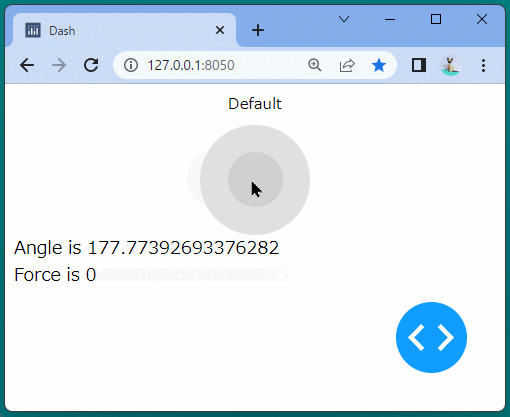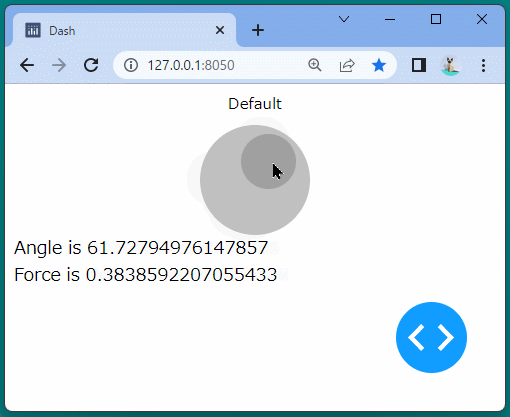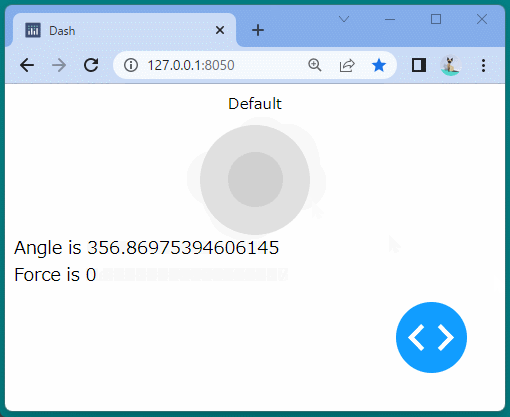
ブール型のインジケーターを表示することができました。
ボタンをクリックするたびに、インジケーターが変化することを確認できます。