DNA塩基配列
DNA塩基配列内の 塩基(A、T、C、G)の出現頻度を分析し、それを円グラフ(パイチャート)で視覚化します。
問題:
与えられたDNA塩基配列から各塩基(A、T、C、G)の出現頻度を計算し、結果を円グラフで表示してください。
解決方法:
- DNA塩基配列を受け取ります。
- 各塩基(A、T、C、G)の出現回数を計算します。
- 出現頻度を円グラフにプロットして視覚化します。
以下はPythonコードの例です:
1 | import matplotlib.pyplot as plt |
このコードでは、DNA塩基配列から各塩基の出現頻度を計算し、それを円グラフで視覚化しています。
各塩基の割合が円グラフ上に表示され、どの塩基が配列内で最も一般的であるかを視覚的に理解できます。
上記のコードを実行すると、DNA塩基の出現頻度が表示された円グラフが生成されます。
ソースコード解説
各ソースコードの部分を説明します。
1. Matplotlibのインポート:
1 | import matplotlib.pyplot as plt |
matplotlib.pyplotモジュールをpltという名前でインポートします。
このモジュールはグラフを作成し、表示するための機能を提供します。japanize_matplotlibライブラリをインポートしています。
これにより、Matplotlibで日本語を正しく表示するためのサポートが追加されます。
2. DNA塩基配列の指定:
1 | dna_sequence = "AGCTAGCTAGCTAGCTAGCTAGCTAGCTAGCTAGCTAGCTAGCTAG" |
- DNAの塩基配列が
dna_sequence変数に格納されています。
3. 各塩基の出現頻度の計算:
1 | a_count = dna_sequence.count('A') |
countメソッドを使用して、各塩基(A、T、C、G)の出現回数を計算しています。
それぞれの結果は個別の変数に格納されます。
4. ラベルと出現頻度のデータの用意:
1 | labels = ['A', 'T', 'C', 'G'] |
- ラベルは各塩基(A、T、C、G)を表し、
labelsリストに格納されています。 - 出現頻度は各塩基の出現回数を示し、
freqリストに格納されています。
5. 円グラフの作成:
1 | plt.figure(figsize=(6, 6)) |
plt.figure関数を使用して、新しい図(グラフ)を作成し、サイズを指定しています。plt.pie関数を使用して円グラフを作成しています。
freqリストがデータとして使用され、labelsリストが各セクターのラベルとして表示されます。autopctパラメータは、各セクター内の割合をパーセンテージで表示するための書式指定を提供します。startangleパラメータは円グラフの始点を角度で指定し、この場合は90度です。
これにより、A塩基から始まるように配置されます。shadowパラメータは、グラフに影を付けるために使用されます。
6. グラフの表示:
1 | plt.show() |
plt.show関数を使用して、作成した円グラフを表示します。
このコードを実行すると、DNA塩基の出現頻度を示す円グラフが生成され、各塩基の割合が視覚的に表示されます。
日本語のテキストが正しく表示されるように、japanize_matplotlib ライブラリが使用されています。
結果解説
円グラフを詳しく説明します。
円グラフは、データの相対的な割合を視覚的に表現するために使用されるグラフの一種です。
DNA塩基の出現頻度を示すために使用されたこの円グラフは、以下の要素で構成されています:
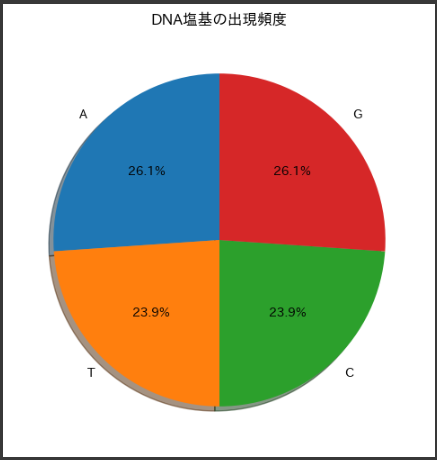
1. ラベル(Labels):
円グラフの中心に各セクター(楔形の部分)に関連付けられたラベルがあります。
この場合、各ラベルはA、T、C、Gとなっています。
これらはDNAの塩基(アデニン、チミン、シトシン、グアニン)を表しています。
2. セクター(Sectors):
円グラフは四つのセクターからなり、各セクターはラベルに対応しています。
セクターの面積は、対応するラベルに関連付けられたデータの割合を表します。
3. 出現頻度(Frequency):
各セクター内の数字は、各DNA塩基の出現回数を表します。
円グラフのセクターは、それぞれの塩基の出現頻度に比例して大きさが異なります。
4. 割合(Percentage):
各セクター内には、そのDNA塩基の全体に対する割合が百分率で表示されています。
これは各塩基の出現頻度を全体に対してどれだけ占めているかを示します。
5. 円グラフのスタイル:
グラフは影を付けて立体感を持たせ、中央から放射状にデータを表現しています。
また、スタートアングルは90度から始まり、反時計回りにデータが表示されています。
この円グラフを解釈すると、DNA塩基の出現頻度がわかります。
各塩基のセクターの面積はその出現頻度に比例しており、円グラフ全体の合計は100%です。
したがって、どのDNA塩基が最も一般的であるか、またそれらの割合がどれだけ異なるかを一目で把握できます。
この具体的な円グラフが示している内容は、与えられたDNA塩基配列内でA、T、C、Gの各塩基がどれだけ出現するか、およびそれらの相対的な割合を示しています。