生物学的ネットワークの解析
NetworkXはPythonのライブラリで、グラフ理論を操作するのに便利です。
生物学的な問題を解決するために、例えば、タンパク質間の相互作用ネットワークや遺伝子の相互作用ネットワークなど、生物学的ネットワークの解析に使用することができます。
以下は、NetworkXを使用してランダムな生物学的ネットワークを作成し、そのネットワークを可視化する例です。
このコードでは、ネットワークのノードはタンパク質や遺伝子を表し、エッジはそれらの間の相互作用を示します。
1 | import networkx as nx |
この例では、ランダムなノードとエッジを持つ生物学的ネットワークを作成して描画しています。
実際の生物学的ネットワークの場合、ネットワーク構造やデータの特性に合わせて異なる手法やアプローチが必要になるかもしれません。
ソースコード解説
このコードは、NetworkXとMatplotlibを使用してランダムな生物学的ネットワークを生成し可視化するものです。
1. ライブラリのインポート:
networkxとmatplotlib.pyplotを使っています。randomモジュールも使われています。
2. ネットワークの生成:
num_nodesで指定された数のノードとnum_edgesで指定された数のエッジを持つ有向グラフを作成します。
まず、空の有向グラフGを作成し、20個のノードをNode_0からNode_19まで追加します。
3. エッジの追加:
num_edgesの数だけランダムなエッジを追加します。
ランダムに2つのノードを選び、それらを結ぶエッジを有向グラフに追加します。
ただし、選ばれた2つのノードが同じでないことを確認して自己ループを避けます。
4. グラフの可視化:
plt.figure()で新しい図を作成し、nx.spring_layout()でノードのレイアウトを設定します。
そして、nx.draw()を使用してノードとエッジを可視化します。
ノードはskyblueの色で、ラベルも表示されます。
エッジはgrayの色で描かれます。
最後にplt.show()でグラフが表示されます。
このコードは、NetworkXを使ってランダムな生物学的ネットワークを生成し、その構造を視覚的に表現しています。
これにより、ネットワーク内のノードとエッジのつながりを視覚化し、ネットワーク全体の特性を把握することができます。
結果解説
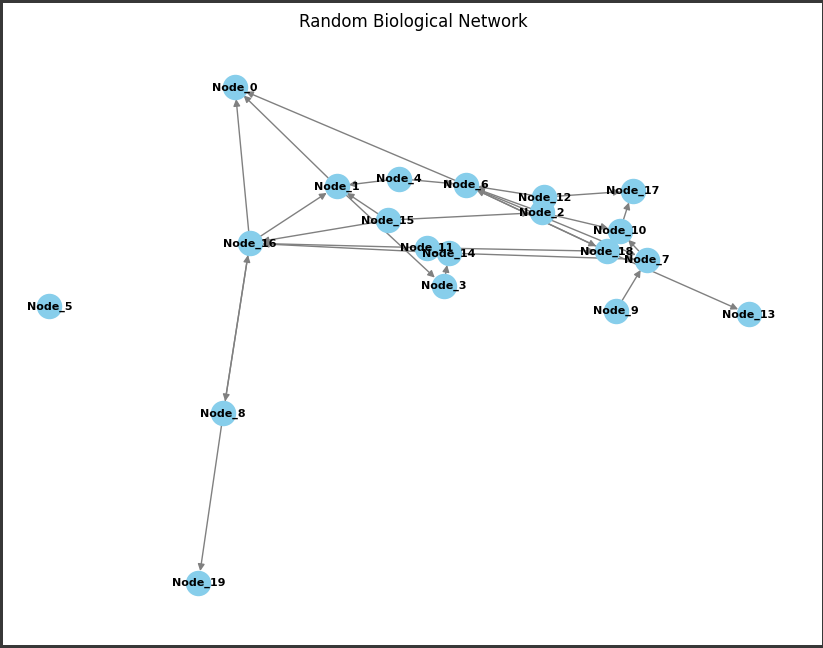
このコードは、NetworkXを使用して作成されたランダムな生物学的ネットワークを可視化しています。
ネットワークは20個のノードと30個のエッジで構成されています。
ノード(Node):
各ノードはタンパク質や遺伝子を表しており、”Node_0”、”Node_1”などの名前で識別されています。
この例では、ネットワーク内のノードは20個生成されています。
エッジ(Edge):
ノード間の相互作用を示しています。
ここでは30個のランダムなエッジがあり、ノード間の結びつきを表しています。
エッジは有向グラフとして表現されており、ノード間の特定の方向性を持っています。
グラフの表示:
nx.spring_layout()を使用してグラフのレイアウトを設定し、ノードの配置を行っています。
ノードのラベルや色、エッジの色などが設定されています。
グラフ全体がランダムな配置で描かれ、ノード同士のつながりが視覚的に表現されています。
このようなネットワークの可視化は、ノード間の関係性やネットワーク全体の特性を理解し、分析するための手助けとなります。
生物学的なネットワークの場合、実際のデータを解析し、そのネットワークの特徴や機能を理解するのに役立ちます。