病状診断
病状診断は医療分野に属する複雑な問題であり、実際の医療診断には十分なデータセットや専門知識が必要です。
ここでは、サンプルとしてシンプルな乳がんの診断を行うコードを示します。
乳がんの診断は二値分類問題で、PyTorchを使用して簡単なニューラルネットワークを構築して実行します。
このサンプルでは、乳がんのデータセットであるBreast Cancer Wisconsin (Diagnostic) Data Setを使用します。
データセットはscikit-learnライブラリから利用できます。
また、結果を分かりやすくするためにMatplotlibを使用してグラフ化します。
まず、必要なライブラリをインポートします。
1 | import torch |
次に、データセットをロードして前処理を行います。
1 | # データセットの読み込み |
次に、簡単なニューラルネットワークモデルを定義します。
1 | class SimpleModel(nn.Module): |
次に、モデルを訓練して、結果をグラフ化します。
1 | # モデルの初期化 |
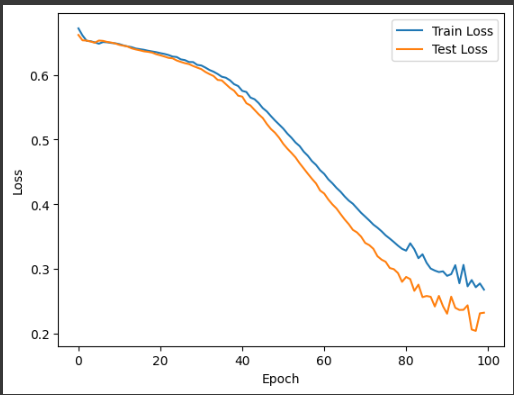
このコードでは、訓練データとテストデータに対する損失の推移を可視化しています。
訓練データとテストデータの損失が共に減少していることが確認できれば、モデルがうまく機能していることが分かります。
ただし、このサンプルはあくまで簡単なものであり、実際の医療診断にはより高度なモデルや専門知識が必要です。
[実行結果]
ソースコード解説
このソースコードは、PyTorchを使用して単純なニューラルネットワークモデルを定義し、乳がんデータセットを使ってそのモデルを訓練し、訓練とテストの損失をプロットするプログラムです。
データの準備:
- ライブラリのインポート: 必要なライブラリをインポートします。
PyTorchのニューラルネットワーク関連のモジュール(torch, torch.nn, torch.optim)やデータセットの読み込みに使うScikit-learn関連のモジュール(sklearn.datasets, sklearn.model_selection)をインポートしています。 - データセットの読み込み: Scikit-learnの
load_breast_cancer()関数を使って乳がんデータセットをロードします。
このデータセットは、乳がんの診断に関連する特徴量と対応するラベル(0: 悪性、1: 良性)を持つものです。
- ライブラリのインポート: 必要なライブラリをインポートします。
データの前処理:
- データをPyTorchのテンソルに変換: NumPy配列からPyTorchのテンソルに変換します。
特徴量Xは浮動小数点数のテンソルに変換され、ラベルyは浮動小数点数のテンソルに変換され、さらに1次元テンソルに変形(.view(-1, 1))されます。 - 訓練データとテストデータに分割:
train_test_split()関数を使ってデータを訓練データとテストデータに分割します。
ここでは、全体の20%をテストデータとして使用し、乱数のシードを固定して再現性を確保しています。
- データをPyTorchのテンソルに変換: NumPy配列からPyTorchのテンソルに変換します。
ニューラルネットワークモデルの定義:
SimpleModelクラス: PyTorchのnn.Moduleを継承した単純なニューラルネットワークモデルを定義しています。
このモデルは、3つの全結合層(nn.Linear)とそれらの間の活性化関数(nn.Sigmoid)で構成されています。
モデルの初期化:
model = SimpleModel():SimpleModelクラスからモデルをインスタンス化します。
損失関数とオプティマイザの設定:
criterion = nn.BCELoss(): 二値交差エントロピー損失関数(Binary Cross Entropy Loss)を使用します。
この損失関数は、二値分類問題に適しています。optimizer = optim.Adam(model.parameters(), lr=0.01): Adamオプティマイザを使用して、モデルのパラメータを最適化します。
学習率(lr)は0.01です。
訓練ループ:
num_epochs = 100: エポック数を100に設定します。
エポックとは、データセット全体を一度学習することを意味します。train_lossesとtest_losses: 訓練中に各エポックの訓練損失とテスト損失を保存するためのリストです。
訓練とテストの実行:
model.train(): モデルを訓練モードに設定します。- 訓練データで順伝播を行い、損失を計算します。
そして逆伝播とオプティマイザのステップ(パラメータ更新)を行います。 model.eval(): モデルを評価モードに設定します。
これにより、ドロップアウトなどの評価時にのみ適用されるレイヤーが適切に動作します。- テストデータで順伝播を行い、テスト損失を計算します。
逆伝播は行いません。
グラフのプロット:
- 訓練とテストの損失をエポックごとにプロットします。
プロットされたグラフは、訓練損失とテスト損失のエポックごとの変化を可視化するのに役立ちます。
- 訓練とテストの損失をエポックごとにプロットします。
このプログラムは、単純なニューラルネットワークを使って乳がんデータセットを分類するタスクを行っており、訓練とテストの損失を監視することでモデルの学習の進行状況を確認できます。